Antibiotika: Bestandsaufnahme zu Präparaten und Unternehmen
Neue Antibiotika gegen Problemkeime werden dringend gebraucht. Einige Pharma-und Biotech-Unternehmen arbeiten daran. Doch noch viel mehr neue Antibiotika werden gebraucht.

Antibiotika – Medikamente gegen schädliche Bakterien – zählen zu den größten Errungenschaften der Medizin. Lungenentzündung, Wundinfektionen, Scharlach, Syphilis und viele weitere Krankheiten haben durch sie ihren Schrecken verloren. Unter den Todesursachen rangieren bakterielle Infektionen deshalb heute in Deutschland weit hinter den Herz-Kreislauf-Krankheiten und Krebs.
vfa-Podcast #MicroScope zu Reserveantibiotika
Download & Streaming
Folgen Sie dem MicroScope-Podcast des vfa kostenfrei über diese Plattformen:
Spotify |Apple Podcasts | Soundcloud | Amazon Music | Deezer | vfa Podcasts

Antibiotika-Klassen und ihre Einführungsjahre
Download des Schaubildes "Antibiotika-Klassen weltweit" als PDF
Mehr als 80 gegen unterschiedliche Bakterienarten wirksame Antibiotika wurden schon entwickelt. Sie gehören verschiedenen Klassen an (siehe Abbildung), die sich jeweils durch eine andere Molekülgrundstruktur und Wirkungsweise auszeichnen. Die meisten neuen Klassen wurden in den 1940er- bis 1960er-Jahren eingeführt; mehrere Klassen aber auch in den 2000er-Jahren, den 2010er-Jahren (hier speziell gegen Tuberkulose) und im Jahr 2025. Die neuen Klassen, die letztes Jahr durch Markteinführung von jeweils einem Antibiotikum in den USA eröffnet wurden, heißen Spiroketal-Topoisomerase-Inhibitoren und Triazaacenaphthylene (gesprochen: Tri-aza-ace-naphthy-lene).
Forschung gegen Antibiotikaresistenzen – Interview mit vfa Präsident Han Steutel
In den 1980er- und 1990er-Jahren
Die meisten Markteinführungen gab es nicht zwischen 1940 und 1969, sondern zwischen 1980 und 1999: Da wurden vor allem die Klassen der Makrolide, Cephalosporine und Fluorchinolonen erweitert (siehe Balkendiagramm). Durch Änderungen in der Molekülstruktur ließen sich die Wirkstoffe beispielsweise so verbessern, dass sie gegen noch mehr Bakterienarten wirksam waren oder die befallenen Gewebe noch besser erreichten.
2001 bis 2010
Nach den 1990er-Jahren änderte sich das Anforderungsprofil für neue Antibiotika radikal. Gut verträgliche Mittel gegen die meisten bakteriellen Infektionen waren ja zahlreich auf dem Markt. Doch zunehmend waren Patienten und Ärzte mit Keimen konfrontiert, die Widerstandskräfte gegen eins oder mehrere dieser Antibiotika entwickelt hatten – sogenannte Resistenzen. Gebraucht wurden daher neue Antibiotika, die gegen diese resistenter Keime wirksam waren, und die meisten der acht in diesem Jahrzehnt neu eingeführten Antibiotika leisteten genau das.
Die Neueinführungen in Deutschland seit 2011
Gram-negative Bakterien
Nur angefärbte Bakterien sind im Lichtmikroskop sichtbar. Mit einer Methode von Christian Gram (1884) lassen sich viele Bakterienarten dunkelviolett anfärben; einige andere allerdings nur schwach rosa. Weil diese Arten die Gram-Färbung so schlecht annehmen, werden sie gramnegativ genannt. Sie haben eine andere Zellwand als andere Bakterien. Das macht sie nicht nur schwerer färbbar, sondern schützt sie auch vor vielen Antibiotika. In den letzten Jahren treten bei einigen von ihnen vermehrt Multiresistenzen auf.
Trotz der Neueinführungen wuchs das Problem der Resistenzen weiter an. Traurige Berühmtheit erlangte der multiresistente "Klinik-Keim" MRSA, ebenso die multiresistente Tuberkulose. Zunehmend wurde auch von Infektionen mit sogenannten multiresistenten Gramnegativen berichtet (einem Typ von Bakterien, gegen den ohnehin nur ein Teil der älteren Antibiotika wirksam ist). Dazu zählt beispielsweise Gonorrhoe (Tripper) durch Neisseria-Bakterien. Einen Überblick über die Resistenzsituation in Deutschland liefert der Bericht GERMAP 2015 der Paul-Ehrlich-Gesellschaft für Chemotherapie und der Abteilung für Infektiologie der Universitätsklinik Freiburg.
Die Antibiotika, die seit 2011 auf den Markt gebracht wurden, sind ausdrücklich gegen Problemkeime entwickelt worden – entweder resistente Formen "gewöhnlicher" Keime oder aber Bakterien, die sich von je her nicht gut haben bekämpfen lassen:
- 2012: Breitband-Antibiotikum Ceftarolin (ein Cephalosporin-Antibiotikum), u. a. gegen den multiresistenten Klinikkeim MRSA wirksam.
- 2013: Antibiotikum Fidaxomicin gegen den Darmkeim Clostridiodes difficile (früher "Clostridium difficile", der Durchfall und schwere Darmkoliken verursacht.
- 2014: Bedaquilin und Delamanid gegen Tuberkulose (die ersten neuen Mittel seit 1995), besiegen in Kombination mit älteren Medikamenten multiresistente Keime besiegen. Dazu kam noch der ältere TB-Wirkstoff Para-Amino-Salicylsäure in neuer, besser einnehmbarer Darreichungsform (Granulat). Mehr dazu hier. Ferner Antibiotikum Telavancin, speziell gegen MRSA bei im Krankenhaus erworbener Lungenentzündung (2018 Marktrücknahme wegen zu geringen Absatzes), sowie das Antibiotikum Ceftobiprol (ein Cephalosporin-Antibiotikum), das gegen Lungenentzündung durch Gram-positive Bakterien (auch MRSA) wie auch nicht-multiresistente gramnegative Bakterien einsetzbar ist.
- 2015: Antibiotikum Tedizolid (ein Oxazolidinon), gegen komplizierte Haut- und Weichteilinfektionen durch Gram-positive Bakterien, auch MRSA
Kombimedikament Ceftolozan + Tazobactam (ein neues Cephalosporin + ein zugelassener Betalactamase-Inhibitor), gegen komplizierte Bauch- und Harnwegsinfektionen mit bestimmten multiresistenten gramnegativen Bakterien - 2016: Antibiotikum Dalbavancin (ein Lipoglycopeptid), gegen komplizierte Hautinfektionen durch grampositive Bakterien, auch MRSA(1)
- 2017: Antibiotikum Ceftazidim + Avibactam (ein Cephalosporin + neuer Betalactamase-Inhibitor) gegen Atemwegs-, Harnwegs- und Bauch-Infektionen durch gramnegative Bakterien (auch solche mit bestimmten Betalactamase-Resistenzen oder mit Klebsiella pneumoniae-Carbapenemase) inkl. Pseudomonas
- 2018: Bezlotoxumab, ein monoklonaler Antikörper, der zusätzlich zu einem Antibiotikum gegen den Darmkeim Clostridiodes difficile angewendet werden kann; er neutralisiert dessen Toxin
- 2019: Antibiotikum Temocillin (ein Penicillin-Derivat) ist gegen einige Resistenzen unempfindlich; in diesem Jahr Markteinführung in Deutschland, zurvor in einigen EU-Ländern schon vertrieben.
- 2021: Antibiotikum Cefiderocol (ein Cephalosporin) zur Anwendung gegen aerobe Gram-negative Bakterien bei begrenzten Therapiemöglichkeiten (da es einige Resistenzen überwindet).
Imipenem / Cilastatin / Relebactam (ein Carbapenem-Antibiotikum mit Booster und neuem Inhibitor von Betalactamasen Typ A und C) zur Anwendung bei im Krankenhaus erworbenen Pneumonien und Infektionen mit gram-negativen Erregern. - 2022: Antibiotikum Eravacyclin (ein Tetracyclin) zur Anwendung bei zahlreichen gram-positiven und gram-negativen Erregern.
- 2024: Antibiotikum Cefepim/Enmetazobactam (ein bekanntes Cephalosporin-Antibiotikum mit einem neuen Betalactamase-Hemmer) zur Anwendung bei zur Behandlung von Erwachsenen mit komplizierten Harnwegsinfektionen (cUTI) oder im Krankenhaus erworbenen Lungenentzündungen (HAP). Antibiotikum Meropenem/Vaborbactam (ein bekanntes Carbabepenem-Antibiotikum mit einem neuen Betalactamase-Hemmer) zur Anwendung bei zur Behandlung von Erwachsenen mit Bauch- oder Harnwegsinfektionen oder im Krankenhaus erworbenen Lungenentzündungen (HAP).
(1) Anders als zwischenzeitlich vom vfa angegeben, wurde das Lipoglycopeptid-Antibiotikum Oritavancin 2016 nicht in Deutschland eingeführt, auch danach nicht. Wir bitten den Fehler zu entschuldigen.
Kommende Antibiotika
Für die Zukunft ist mit einigen weiteren Antibiotika zu rechnen. Einen Überblick darüber gibt die Seite antibakterielle Pipeline. Auch wenn ihre Zahl auf den ersten Blick nicht gering erscheint, werden doch noch viel mehr Antibiotika gebraucht, weil sie a) unterschiedliche Resistenzen überwinden, weil es b) nur ein Teil davon zur Zulassung schaffen wird, und weil c) auch diese Medikamente vermutlich nicht dauerhaft vor Resistenzen verschont bleiben.
Antibiotika-Entwicklung in Unternehmen
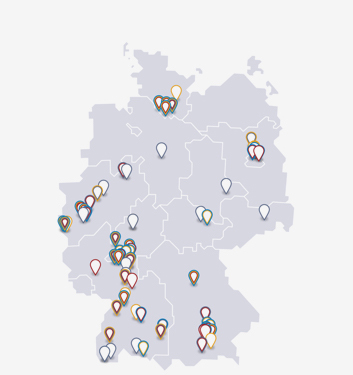
Derzeit arbeiten weltweit ein paar große, vor allem aber mehr als 50 kleine und mittlere Unternehmen an neuen Antibiotika und anderen antibakteriell wirksamen Medikamenten.
Pharma- und Biotech-Unternehmen, die neue Antibiotika und andere antibakterielle Medikamente entwickeln (nach vfa-Recherchen, Stand 01.11.2022)
| Große Unternehmen: | Kleine und mittlere Unternehmen: |
| GSK (UK, Labore in USA), Janssen (USA), Menarini (I; klinische Entwicklung), MSD (USA), Otsuka (JP), Pfizer (USA; keine eigene Wirkstoff-Forschung), Roche (CH), Shionogi (JP) | AiCuris (D), Allecra Therapeutics (D), Basilea Pharmaceutica (CH), BC World (S-Korea), Boston Pharmaceuticals (USA), Cardeas Pharma(2) (USA), cellceutix (USA), Cempra (USA), Cosmo Pharmaceuticals (I), CrystalGenomics (S-Korea), CURx Pharmaceuticals (USA)(2) , Cyclenium (Kanada), Debiopharm (CH), Destiny Pharma (UK), Discuva (UK), Dong-A (S-Korea), Dream Pharma (S-Korea), Durata Therapeutics (USA), Elorac (USA), Enanta (USA), Entasis (USA), Evotec (D), Infectex (Russland), Juvabis (CH), Kyorin (JP), Lee’s Pharmaceutical(2) (Hong Kong), LegoChem Biosciences (S-Korea), Lisando (Liechtenstein, Labore in D), Lytix Biopharma (N), Macrolide (USA), Melinta (USA), MerLion Pharmaceuticals (Singapur), MicuRx Pharmaceuticals (USA und China), Nabriva (AU), Naicons (I), Nanotherapeutics (USA)(2) , NovaBay Pharmaceuticals (USA), Orchid Pharmaceuticals (Indien), Paratek Pharmaceuticals (USA), RedHill Biopharma (Israel)(2) , Sequella (USA), Sihuan Pharm (China), Spexis (CH), Summit (UK), Tetraphase Pharmaceuticals (USA), The Medicines Company (USA), Theravance Biopharma (USA), VenatorRx (USA), Wockhardt (Indien und USA), Zavante Therapeutics (USA) u. a. |
Die meisten davon betreiben ihre Labors in den USA, doch auch in Deutschland, Österreich und der Schweiz arbeiten industrielle Pharma-Forscher an neuen Antibiotika für die Humanmedizin. Das zeigt die folgende Karte.